Autour des données
Présentation du chapitre
Dans ce chapitre nous allons cheminer à travers diverses techniques et représentations de la donnée structurée (tableaux de classes et de chiffres) en complément à ce que nous avons déjà abordé.
Précédemment, en ce qui concerne l’interprétation des données, le point de vue développé était celui de la préparation des données avant leur usage dans un algorithme supervisé ou non, en grande partie pour les rendre ingérables par cet algorithme. Ici, à la demande de nos lecteurs, nous nous intéressons à la manipulation de la donnée en soi, à la fois pour la préparer, mais aussi pour être à même d’émettre de premières hypothèses ou d’avoir de premières intuitions sur sa signification. Évidemment, les deux points de vue sont complémentaires !
L’agencement de ce chapitre ne doit pas être interprété comme un "protocole" ou une méthode d’analyse de la donnée. Nous avons fait un choix didactique qui introduit pas à pas divers outils, ou qui présente la même chose sous divers angles, afin que le lecteur dispose d’éléments complémentaires à ce qu’il sait déjà, pour qu’il...
Appréhension directe et rapide d’un dataset
Afin d’éviter des redondances inutiles, nous allons nous doter d’un dataset dont les données seront utilisées dans le chapitre qui traite de la création d’un modèle sans programmation via un outil comme le logiciel BigML. Le package mlbench permet d’accéder à ces données et également de fabriquer des datasets adaptés à un type de problème.
library(tidyverse)
library(mlbench) # package pour créer ou accéder à des données de test On peut facilement lister l’ensemble des datasets de démonstration proposés dans les packages qui sont installés sur une machine.
data(package = .packages(all.available = TRUE)) # liste des datasets disponibles Voici un (très) court extrait de ce qui est disponible sur notre machine :
...
Data sets in package ‘mlbench':
BostonHousing Boston Housing Data
BreastCancer Wisconsin Breast Cancer Database
DNA Primate splice-junction gene sequences (DNA)
Glass Glass Identification Database
HouseVotes84 United States Congressional Voting Records 1984
Ionosphere Johns Hopkins University Ionosphere database
LetterRecognition Letter Image Recognition Data
Ozone Los Angeles ozone pollution data, 1976
PimaIndiansDiabetes Pima Indians Diabetes Database
... Nous allons invoquer les données suivantes :
data(PimaIndiansDiabetes) # accès aux données diabète...Analyse de la conformation des distributions par rapport à la loi normale
On peut valider certaines de nos intuitions par rapport à la normalité des données en confrontant ces densités à la loi normale en utilisant un diagramme Quantile-Quantile.
# ajustements loi normale(mu,e)
plot_qq(df) # diagrammes Quantile-Quantile 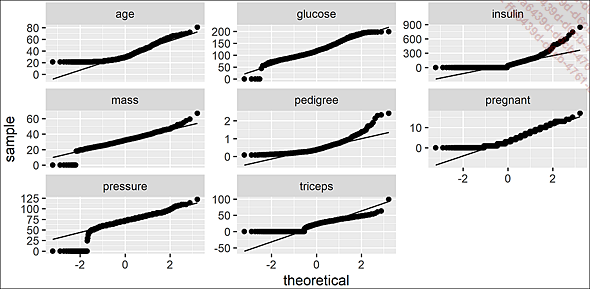
Diagrammes Quantile-Quantile
Même sans savoir interpréter finement un tel diagramme, cela attire notre attention sur certaines caractéristiques des données :
-
Il faudra analyser les valeurs nulles (nulles ou inconnues ?) au sein de glucose, insulin, mass, pregnant, pressure, triceps, ce qui nous permettra de décider notre stratégie (remplacement par NA, par une moyenne, suppression de lignes de mauvaise qualité, traitement particulier d’outliers…).
-
Le fait que glucose, mass et pressure ressemblent à des droites proches de la diagonale (hors valeurs nulles à qualifier) peut nous laisser penser que, conformément à leur aspect général dans la figure Densités, par variable, elles sont plus ou moins approximables par une loi normale.
-
La forme régulière de pedigree pourrait nous inciter à la rapprocher d’une autre loi (à tester), typiquement une loi de Poisson ou Log-normal.
On peut comparer cet ajustement en distinguant...
Dépendance linéaire entre variables
En complément, il est d’usage de vérifier si certaines des variables explicatives ne sont pas en forte corrélation, ce qui pourrait inciter à essayer des modèles de prédiction qui ne garderaient que l’une ou l’autre de ces variables (pour bien faire, on compare l’efficacité avec l’une et avec l’autre, et quand elle est similaire, on conserve la variable la plus facile à interpréter ou à collecter dans le monde réel).
plot_correlation(df) # un tableau de corrélations 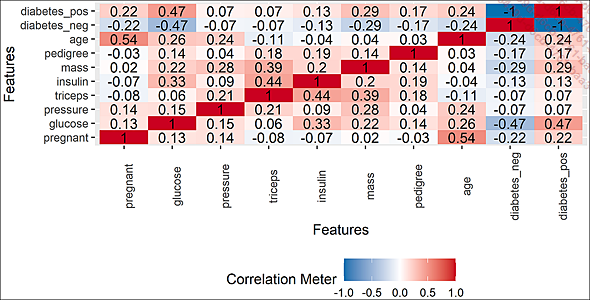
Corrélations deux à deux
Notre problème n’est pas linéairement trivial, le diabète n’est manifestement pas corrélé simplement à une variable. Excepté pour la variable age, il semble totalement exclu qu’il y ait une part de relation plus ou moins linéaire et simple entre le nombre de grossesses et les autres variables explicatives. On peut se représenter cela, peut-être à tort, en imaginant qu’il serait intéressant de réfléchir à imaginer deux grandes familles de prédicteurs : prédicteurs "sociaux" (âge, nombre de grossesses) et prédicteurs techniques (les autres). Tout cela étant posé, remarquez que cette piste ne porte que sur des relations linéaires...
Mise en évidence de différences sur les distributions
Nous allons maintenant tâcher de mettre en évidence (ou non) d’éventuelles différences de distributions pour certaines variables d’aspect gaussien (pour cet exemple, nous avons choisi glucose) entre la distribution avec diabète ou non.
Notre préoccupation principale sera la significativité de l’éventuelle différence de moyenne. L’outil nous donne également des informations sur la différence d’écart-type, qui ne nécessite pas d’interprétation forte, sinon que le fait qu’elle soit significative peut nous conforter dans l’idée qu’un algorithme judicieux saura en extraire des améliorations de prédictibilité du diabète.
library(lessR)
d <- df # par défaut lessR utilise d comme dataset
tt.brief(glucose ~ diabetes) # graphique et stats 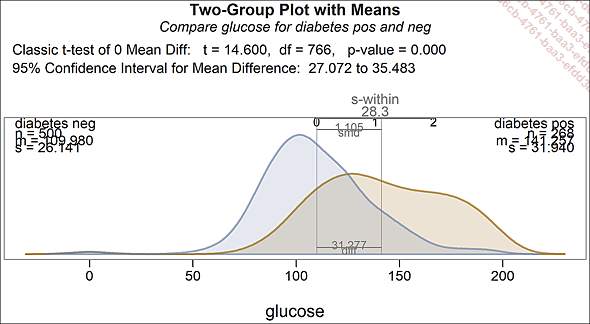
Distributions du glucose en fonction du diabète
La différence de moyenne est significative !
Vous pourriez aussi vouloir une représentation en "moustache/violon", en utilisant la syntaxe suivante :
library(ggstatsplot)
set.seed(666) ...Points étrangers
Nous avons déjà travaillé sur l’identification d’outliers. La représentation suivante va permettre d’extraire des observations candidates à ce que vous les qualifiiez comme points étrangers. C’est à vous de choisir par essais successifs de différents paramètres le pouvoir plus ou moins discriminant du contour que vous allez tracer. Il vous reste à étudier individuellement les points à l’extérieur, dont l’identifiant est stipulé, pour commencer à envisager votre stratégie de traitement de ce type de point.
Le fait de conserver des points techniquement aberrants (anomalies ou pas) peut être valide ou non, suivant le traitement que vous appliquez sur ces valeurs. Le fait de conserver ou pas des points correspondant à des observations exceptionnelles peut vous aider à diminuer l’overfitting ou, à l’inverse, diminuer la prédictibilité de vos modèles. Toute décision de ce type doit être soigneusement consignée comme hypothèse structurante dans la documentation de vos modèles. Vous serez souvent amené à challenger ces hypothèses et à faire évoluer vos positions quant à celles-ci pour améliorer votre travail. Ne prenez pas pour argent comptant les assertions que vous pouvez...
Tris et agrégats
1. Tris automatisés
Nous allons maintenant aborder différents tris (dont le tri à plat) de nos données au travers de différentes fonctions spécialisées.
Pour nous aider dans la présentation de nos résultats, nous avons rédigé une fonction naïve de présentation de ceux-ci, qui nous servira à plusieurs reprises dans cette section :
# une petite fonction pour imprimer head + tail d'un objet data
h_t <- function(x, titre = "", sous_titre ="", n = 3)
{
cat(titre, "\n")
cat(sous_titre, "\n")
print(rbind(head(x,n),tail(x,n)))
} Nous nous proposons de visualiser les répartitions d’effectifs entre le nombre de grossesses (qui était le résultat d’un comptage, et donc une variable quantitative ordonnée) et le diabète. Le tri le plus simple s’effectue comme suit en utilisant l’instruction R table :
# tri à plat sur 2 var discrètes
t <- table(df$pregnant,df$diabetes) # tri simpliste
h_t(t,
"6 lignes extraites d'un simple tri à plat, nb grossesses vs statut diabète",
"----------"
)
6 lignes extraites d'un simple tri à plat, nb grossesses vs statut diabète
----------
neg pos
0 73 38
1 106 29
2 84 19
14 0 2
15 0 1
17 0 1 Ce comptage peut être utile, en particulier si vous créez un dataset réutilisable, mais vous voudrez sans doute l’enrichir avec des sommes. Notez que addmargins vous permet de choisir une autre fonction que la somme.
t <- addmargins(table(df$pregnant,df$diabetes)) # tri avec les sommes 6 lignes, tri à plat avec sommation, nb grossesses vs statut diabète
---------------
neg pos Sum
0 73 38 111
1 106 29 135
2 84 19 103
15 0... Editions ENI Livres | Vidéos | e-Formations
Editions ENI Livres | Vidéos | e-Formations